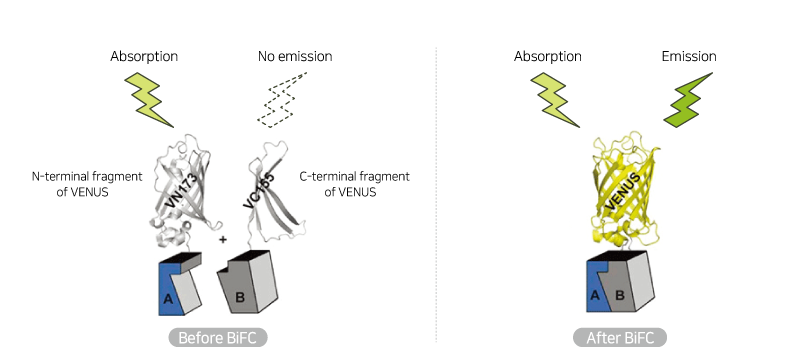
VENUS는 생물학 연구에 일반적으로 사용되는 녹색 형광 단백질(GFP)의 노란색 변종 (YFP)에서 파생된 형광 단백질입니다. VENUS는 GFP에 비해 더 밝은 노란색 형광 신호를 방출하므로 세포에서 더 쉽게 감지되며, 또한 빠르게 접히고 성숙이 빨라 단백질 위치를 조기에 시각화 할 수 있습니다. 또한 VENUS는 광독성이 낮아 영상 실험 중 빛에 장기간 노출되어 발생하는 세포 손상을 최소화합니다. 일반적으로 VENUS protein은 효모 세포의 단백질 위치, 발현 및 상호작용 연구하는데 활용될 수 있습니다.
- 세포 내 위치: 살아있는 세포에서 형광 VENUS 신호를 관찰함으로써 연구자들은 VENUS에 융합된 표적단백질의 세포 위치를 확인할 수 있습니다. 이는 단백질이 세포 내에서 어디에 기능하는지 이해하는 데 도움이 됩니다.
- 발현: VENUS 신호의 강도는 세포 내 표적단백질의 발현 수준을 나타내는 지표가 될 수 있습니다.
- 단백질-단백질 상호 작용: 서로 다른 VENUS 태그가 붙은 단백질을 공동 발현하는 균주를 생성함으로써 연구자들은 BiFC (이분자 형광 상보기법)와 같은 기술을 활용하여 서로 상호 작용하는 단백질을 식별할 수 있습니다. 상호작용하는 VENUS 단편이 붙은 두 단백질이 가까이 모이면, VENUS 단편은 완전한 형광 단백질을 형성할 수 있을 만큼 가까워져 신호를 생성합니다.
바이오니아는 VENUS 형광 단백질과 표적단백질이 결합한 융합 단백질을 발현하는 haploid S.cerevisiae를 제작하였으며, S.cerevisiae Genome-wide VENUS-Fusion Library로 제공하고 있습니다. VENUS 단백질은 N-말단 단편 (VN) 혹은 C-말단 단편 (VC)의 형태로 표적단백질과 결합되어 있으며, VN과 VC 단편이 결합하여 완전한 VENUS 단백질 구조가 되면 형광을 나타냅니다. 그리고 이 VENUS-표적단백질 형태의 융합단백질은 표적유전자 프로모터 조절하에 발현됩니다. S.cerevisiae Genome-wide VENUS-Fusion Library는 VC-와 VN-fusion library로 제공되고 있습니다.
- S.cerevisiae VC-fusion library (5,552 strains): C-말단 단편의 VENUS 형광 단백질 (VC)이 표적단백질에 결합되어 있으며, MATa (mating type)의 haploid S.cerevisiae 균주에 제작되었습니다.
- S.cerevisiae VN-fusion library (5,809 strains): N-말단 단편의 VENUS 형광 단백질 (VN)이 표적단백질에 결합되어 있으며, MATα (mating type)의 haploid S.cerevisiae 균주에 제작되었습니다.
이배체 효모 (diploid yeast)와 반수체 효모 (haploid yeast)는 가지고 있는 염색체 수에 차이가 있습니다.
- 이배체 효모 (diploid yeast): 이배체 효모 세포에는 두 세트의 염색체 (2n)가 있습니다. 이는 일반적으로 각 염색체에 대해 두 개의 복사본이 있고 각 부모로부터 하나씩 물려받은 것을 의미합니다.
- 반수체 효모 (haploid yeast): 반수체 효모 세포에는 단일 세트의 염색체 (n)가 있습니다. 반수체 효모 세포는 일반적으로 교배 사건이나 특정 유전적 조작을 통해 발생합니다. 반수체 효모는 교배 유형 (mating type)에 따라 유성생식을 통해 이배체 효모로 만들어질 수 있습니다.
빵 효모로도 알려져 있는 Saccharomyces cerevisiae는 유성생식을 하며 MATa와 MATα 두 가지 교배 유형이 있습니다. 이러한 교배 유형은 3번 염색체에 위치한 단일 교배 유형 유전자좌 (MAT 유전자좌)에 의해 결정됩니다.
- MATa 유형: 이 유형은 MAT 유전자좌에 MATa1과 MATa2 유전자 서열을 포함합니다.
- MATα 유형: 이 유형은 MAT 유전자좌에 MATα1과 MATα2 유전자 서열을 포함합니다.
다른 교배유형을 갖는 S.cerevisiae는 아래와 같은 방식으로 유성생식을 합니다.
- 효모세포 접촉: 반대 교배 유형의 반수체 세포 (MATa 및 MATα)는 먼저 응집이라는 과정을 통해 물리적 접촉을 거쳐야 합니다.
- 신호 전달: 각 교배 유형에 따라 페로몬이 분비됩니다. MATa 세포는 a-인자라고 불리는 페로몬을 분비하고, MATα 세포는 α-인자를 분비합니다. 이 페로몬은 반대 교배 유형의 특정 수용체에 결합하여 신호 전달 계통을 촉발합니다.
- 세포 융합: 페로몬 신호 전달 경로는 세포벽 리모델링과 두 세포 사이의 융합 다리 형성을 유도합니다.
- 핵융합: 세포벽은 궁극적으로 완전히 파괴되어 두 개의 반수체 핵이 융합되어 이배체 세포를 형성합니다.
그리고 교배결과로 생성된 이배체 효모 세포는 각 부모로부터 염색체를 하나씩 받아 두 세트의 염색체를 갖게 됩니다. 이러한 반수체 효모 교배기술은 유전학 연구, 포자 분석, 발효균주 최적화 연구 등에 사용될 수 있습니다.
이분자 형광 상보기법 (BiFC, bimolecular fluorescence complementation)은 살아있는 세포에서 단백질-단백질 상호작용 (PPI, protein-protein interaction)을 연구하는 데 사용되는 기술입니다. S.cerevisiae Genome-wide VENUS-Fusion Library를 활용한 BiFC assay 원리와 방법은 아래와 같습니다.
- VENUS fusion library는 S.cerevisiae VC-fusion library와 S.cerevisiae VN-fusion library로 구분되어 있습니다. VC-fusion library는 C-말단 단편의 VENUS 형광 단백질 (VC)이 표적단백질에 결합되어 있으며, VN-fusion library는 N-말단 단편의 VENUS 형광 단백질 (VN)이 표적단백질에 결합되어 있습니다. 두 라이브러리 모두 haploid S.cerevisiae를 활용하여 제작되었지만, 교배유형은 각각 MATα (VC fusion library)와 MATa (VN fusion library)로 다릅니다.
- 상호작용을 확인하고 싶은 두 종류의 표적단백질 A와 B를 선정한 후 동일하지 않은 VENUS 말단과 결합된 표적단백질 A와 B 발현 균주를 선택합니다. 예를 들면, VC-fusion library에서 VC-protein A 발현균주와 VN-library에서 VN-protein B 발현균주를 선택합니다.
- VC- 및 VN-fusion library S.cerevisiae 교배유형이 각각 MATα와 MATa로 다르기 때문에 유성생식을 교배가 가능합니다. 따라서 VC-와 VN-fusion library 균주 교배를 수행합니다. 이때 만들어진 이배체 효모 세포에는 각각 C-말단과 N-말단 단편의 VENUS 형광단백질과 결합된 표적단백질 A, B 모두 존재하게 됩니다.
- 단백질 A와 단백질 B가 자연적으로 가까워지며 상호작용이 충분히 강하면, 같이 결합되어 있던 VN과 VC 단편이 재조립되어 완전한 형광 VENUS 단백질을 형성하게 됩니다. 이렇게 재조립된 VENUS는 노란색 형광 신호를 방출하는데, 이는 단백질 A와 단백질 B 사이의 잠재적인 상호작용을 나타냅니다. 반대로 만약 두 단백질이 상호작용하더라도 상호작용이 약하거나 일시적이라면, VN과 VC 단편이 충분히 가까워지지 않아 형광을 나타내지 않을 수 있습니다.
S.cerevisiae는 30℃의 온도에서 생육하며, 주요 배지 조성은 아래와 같습니다.
1. YPD for general culture and maintenance medium
| Component |
Amt |
Final conc. |
| Yeast extract |
10 g/L |
1.0% w/v |
| Peptone |
20 g/L |
2.0% w/v |
| Glucose |
20 g/L |
2.0% w/v |
| Solid media is made by adding 2% Bacto Agar |
2. SC medium for auxotrophic marker selection
- SC-Ura (S.cerevisiae VN-Fusion library selection): SD 배지에 various additions (Ade, Leu, Trp, His, Arg, Met, Tyr, Ile, Lys, Phe, Glu, Asp, Val, Thr, Ser)를 첨가합니다.
- SD-Leu (S.cerevisiae VC-Fusion library selection): SD 배지에 various additions (Ade, Ura, Trp, His, Arg, Met, Tyr, Ile, Lys, Phe, Glu, Asp, Val, Thr, Ser)를 첨가합니다.
- SD medium (Synthetic minimal glucose medium)
| Component |
Amt |
Final conc. |
| Bacto-yeast nitrogen base (w/o a.a) |
6.7 g/L |
0.67% w/v |
| Glucose |
20 g/L |
2.0% w/v |
| Solid media is made by adding 2% Bacto Agar |
- Various additions for SC medium
| Constituent |
Final conc. (mg/L) |
Stock (g) per 100 ml |
Amt. of stock (ml) per L |
| Adenine sulfate |
20 |
0.2 |
10 |
| Uracil |
20 |
0.2 |
10 |
| L-Tryptophan |
20 |
1 |
2 |
| L-Histidine-HCl |
20 |
1 |
2 |
| L-Arginine-HCl |
20 |
1 |
2 |
| L-Methionine |
20 |
1 |
2 |
| L-Tyrosine |
30 |
0.2 |
15 |
| L-Leucine |
60 |
1 |
6 |
| L-Isoleucine |
30 |
1 |
3 |
| L-Lysine-HCl |
30 |
1 |
3 |
| L-Phenylalamine |
50 |
1 |
5 |
| L-Glutamic acid |
100 |
1 |
10 |
| L-Aspartic acid |
100 |
1 |
10 |
| L-Valine |
150 |
3 |
5 |
| L-Threonine |
200 |
4 |
5 |
| L-Serine |
400 |
8 |
5 |
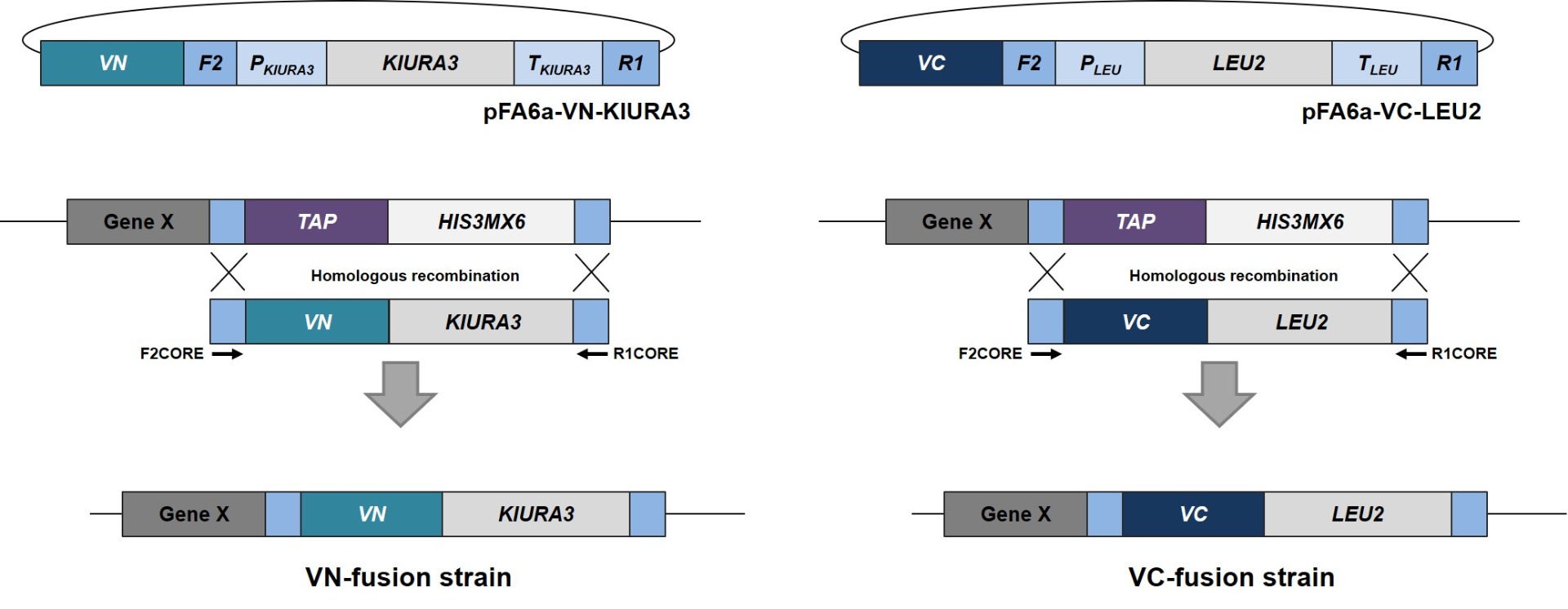
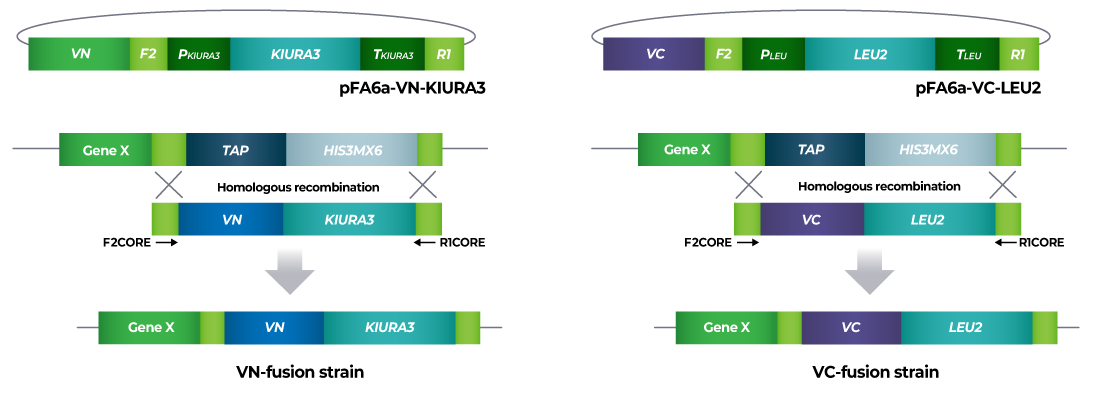
S.cerevisiae Genome-wide VENUS-Fusion Library는 표적유전자에 VENUS 단백질의 N-말단 단편 (VN) 혹은 C-말단 단편 (VC)이 결합된 융합단백질을 발현하는 S.cerevisiae 모음입니다. S.cerevisiae Genome-wide VENUS-Fusion Library는 표적단백질에 결합된 VENUS 단편에 따라 VC-fusion library와 VN-fusion library로 구성됩니다. 두 라이브러리 모두 S.cerevisiae TAP-tagged collection를 이용하였으며, 상동 유전자 재조합 방법을 통해 DNA 카세트를 삽입하는 방식으로 제작되었습니다.
1. 표적단백질 선택
VC 혹은 VN과 결합할 표적단백질을 선택하고, S.cerevisiae TAP-tagged collection에서 관련 균주를 확인합니다.
2. DNA 카세트 준비
상동성 재조합 (homologous recombination)으로 VC 혹은 VN을 표적단백질에 결합한 융합단백질을 발현하는 효모 균주를 제작하기 위한 DNA 카세트를 준비합니다. DNA 카세트의 양쪽 측면영역에는 TAP-tagged collection 제작에 사용된 F2CORE와 R1CORE 서열이 존재하며, 그 사이에는 VC 혹은 VN 서열과 효모의 선택적 선발을 위한 영양요구성 마커 유전자가 삽입되어 있습니다. 영양요구성 마커로 VC-fusion library에서는 KIURA3 (uracil), VN-fusion library에는 LEU2 (leucine) 유전자가 활용되었습니다.
3. 효모 형질전환
형질전환 기술을 이용하여 DNA 카세트를 효모 세포에 도입합니다. 효모 세포에 들어간 DNA 카세트는 게놈상의 표적유전자 근처에 위치한 F2CORE와 R1CORE 서열을 인식한 후 해당 위치로 삽입됩니다. 이때 TAP-tagged collection에 존재하던 TAP-HISMX6는 효모게놈에서 사라지며, 대신 DNA 카세트의 VENUS 단백질 단편과 영양요구성 마커으로 대체됩니다.
4. VENUS fusion 균주 선발
형질전환된 효모는 영양요구성 배지에 생장시켜 DNA 카세트가 게놈에 도입된 효모만을 선발합니다. 마지막으로 모든 VENUS fusion 균주를 하나의 세트로 준비하여 라이브러리로 제공합니다. 선발된 효모에서 표적단백질은 VC 혹은 VN과 결합된 형태로 발현되며, 표적단백질 유전자의 자체 프로모터에 의해 발현이 조절됩니다.
Agar type와 glycerol type은 배양된 효모의 보관 조건입니다.
Agar (한천) type: 효모 균주를 미리 제조된 영양분이 풍부한 고체배지에 배양하여 보관하는 방식으로 agar는 액체형태의 배지를 고형화 시키는 역할을 합니다. Agar type의 균주는 30℃에서 배양 후 생장을 억제하기 위해 4℃에 보관합니다.
Glycerol (글리세롤) type: 액체배양된 균주에 glycerol를 30% (v/v)로 처리하여 동결보존 (-70℃) 할 수 있도록 하는 보관 조건입니다. Glycerol은 세포 내 물분자와 결합하여 낮은 온도에서도 얼음결정 생성을 억제하여 cell을 보호하는 cryoprotectant 역할을 합니다.
바이오니아에서 효모균주 주문 시, shipping type으로 아래와 같이 agar type 및 glycerol type 중 선택하실 수 있습니다.
- Agar type: Rich medium + supplements solid medium (2.0 microtube)
- Glycerol type: Rich medium + supplements + 30% glycerol liquid medium (2.0 microtube)
| Cat. No. |
Pack size |
Shipping temperature |
| 가로 (cm) |
세로 (cm) |
길이 (cm) |
제품무게 (kg) |
드라이아이스 포함무게 (kg) |
총 무게 (kg) |
| M-1010 (Agar type) |
22.5 |
16.5 |
5.8 |
0.158 |
- |
0.158 |
상온 |
| M-1010 (Glycerol type) |
5.4 |
4.5 |
5 |
0.049 |
10 |
10.049 |
냉동 |
*Glycerol type은 냉동 배송으로 dry ice비용이 추가됩니다.
S.cerevisiae 게놈 데이터베이스는 대표적으로 Saccharomyces Genome Database (SGD, https://www.yeastgenome.org)에서 확인하실 수 있습니다. SGD에서는 S.cerevisiae의 분자생물학 및 유전학에 대한 포괄적인 정보를 무료로 제공합니다. 그 외에 Ensembl Fungi S.cerevisiae (https://fungi.ensembl.org/Saccharomyces_cerevisiae/Info/Index)와 NCBI (https://www.ncbi.nlm.nih.gov/datasets/genome/)에서도 정보를 얻으실 수 있습니다.
S.cerevisiae 유전자의 human ortholog gene을 찾는 데 두 가지 주요 방법이 있습니다.
1. SGD 사용
대표적인 S.cerevisiae 게놈 데이터베이스인 SGD는 human ortholog gene 찾는 데 필요한 리소스를 제공합니다. 사용 방법은 아래와 같습니다.
- SGD에서 관심 있는 S.cerevisiae 유전자의 유전자 페이지로 이동합니다. 검색창을 이용하여 이름이나 유전자 ID로 유전자를 검색할 수 있습니다.
- 결과페이지 우측 상단의 "Homology" 섹션을 클릭합니다. 페이지 중단에 위치한 “Homologs”에서 Filter table에 “Homo sapiens”를 입력하시면, 해당 유전자의 human ortholog gene을 확인하실 수 있습니다.
2. 서열 유사성 기반 탐색
S.cerevisiae 유전자에 대해 human ortholog gene을 찾을 수 없는 경우, BLAST (Basic Local Alignment Search Tool)와 같은 서열 유사성 검색 도구를 활용할 수 있습니다. 대표적인 NCBI BLAST 사용방법은 아래와 같습니다.
- 관심있는 S.cerevisiae 유전자의 DNA 혹은 단백질 서열을 알아야 합니다. 이 정보는 연구를 통해 확보하거나 SGD 혹은 NCBI 데이터베이스 (https://www.ncbi.nlm.nih.gov/search/)에서 유전자 ID를 사용하여 확인할 수 있습니다.
- 다음 NCBI BLAST 페이지에 접속한 후 (https://blast.ncbi.nlm.nih.gov/Blast.cgi), 보유한 DNA 서열 (nucleotide)이나 단백질 (protein) 서열을 활용할 수 있도록 blastn, blastp 혹은 blastx를 선택합니다. 그리고 “Organism” 탭에서 “homo sapiens”를 선택한 후 화면 아래의 BLAST를 클릭합니다.
- BLAST 결과에서 S.cerevisiae 서열과 가장 서열 유사성이 높은 human ortholog 서열이 가장 낮은 E-value와 함께 결과 최상단에 위치합니다.