|
Genome-wide evidences of bisphenol a toxicity using Schizosaccharomyces pombe
Dong-Myung Kim, Jeonghoon Heo, Dong Woo Lee, Mayumi Tsuji, Mihi Yang
Archive of Pharmacal Research. 2018 Aug;41(8):830-837. • More information
Identification of a Mitochondrial DNA Polymerase Affecting Cardiotoxicity of Sunitinib Using a Genome-Wide Screening on S. pombe Deletion Library
Dong-Myung Kim, Hanna Kim, Ji-Hyun Yeon, Ju-Hee Lee, Han-Oh Park
Toxicological Sciences. 2016 Jan;149(1): 4–14. • More information
The Natural Anticancer Agent Plumbagin Induces Potent Cytotoxicity in MCF-7 Human Breast Cancer Cells by Inhibiting a PI-5 Kinase for ROS Generation
Ju-Hee Lee., Ji-Hyun Yeon., Hanna Kim, Whijae Roh, Jeiwook Chae, Han-Oh Park, Dong-Myung Kim
PLoS ONE. 2012 Sep;7(9): e45023. • More information
Analysis of a genome-wide set of gene deletions in the fission yeast Schizosaccharomyces pombe
Dong-Uk Kim, Jacqueline Hayles, Dongsup Kim, Valerie Wood, Han-Oh Park, Misun Won, Hyang-Sook Yoo, Trevor Duhig, Miyoung Nam, Georgia Palmer, Sangjo Han, Linda Jeffery, Seung-Tae Baek, Hyemi Lee, Young Sam Shim, Minho Lee, Lila Kim, Kyung-Sun Heo, Eun Joo Noh, Ah-Reum Lee, Young-Joo Jang, Kyung-Sook Chung, Shin-Jung Choi, Jo-Young Park, Youngwoo Park, Hwan Mook Kim, Song-Kyu Park, Hae-Joon Park, Eun-Jung Kang, Hyong Bai Kim, Hyun-Sam Kang, Hee-Moon Park, Kyunghoon Kim, Kiwon Song, Kyung Bin Song, Paul Nurse, Kwang-Lae Hoe
Nature Biotechnology. 2010 Jun;28(6): 617-623. • More information
|







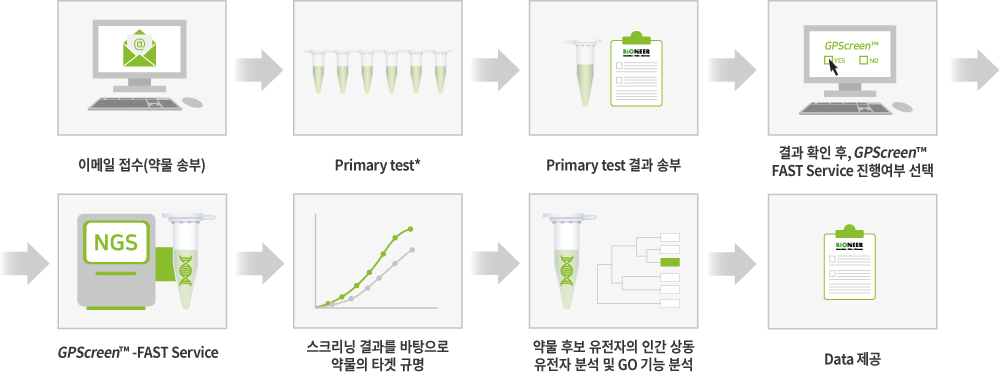

▶ GPScreen을 통해 발굴된 약물 작용점을 이용하여 인체 세포에서 타겟 검증 연구가 가능합니다.
I인체세포에서의 약물 타겟 검증 방법
바이오니아는 약 2만여 종의 인체 유전자에 대한 siRNA 라이브러리를 보유하고 있으며,
발굴된 타겟의 human ortholog를 이용하여 인체 세포에서의 유전자 발현 억제를 통한 타겟을 검증할 수 있습니다.
1) 인체 세포에서 약물에 의한 인체 상동 유전자 발현 조절 분석
Figure 1. Sunitinib 이 Hela 인간 세포주에서 인간 상동 유전자인 POLG의 발현을 억제시키는 것을 확인
(A) 인체 세포에서의 sunitinib 효과를 확인하기 위해 인간 자궁경부암 세포주인 Hela 세포에 sunitinib을 처리한 후 POLG 단백질의 발현량을 확인했습니다. POLG 항체와 goat anti-rabbit IgG-TRITC 항체를 이용하여 세포면역염색 후 컨포컬 현광현미경에서 관찰했습니다.
(B) Hela 세포에 sunitinib을 처리한 후 POLG mRNA 양을 qRT-PCR로 측정했습니다.
2) 바이오니아의 human siRNA 라이브러리를 이용하여 인간상동유전자 발현 억제 효과 분석
Figure 2. 인간 자궁경부암 세포주인 Hela 세포에서 POLG 발현 억제 시 sunitinib 에 의한 세포 독성이 현격히 증가
(upper panel) Hela 세포주에 POLG 타겟 siRNA(5 nM, 48h)를 처리한 후 세포에 sunitinib (5 µM, 48h) 을 처리하여 SRB assay로 세포 양을 측정하였습니다.
(bottom panel) SRB assay 한 세포를 광학 현미경으로 관찰하였습니다.
IIGPScreen™ 기술의 신약개발 후보 도출에의 적용
추가로, GPScreen™ 기술은 신약 개발 초기 단계에서 약물 후보들 간의 우수 화합물 선별에도 이용 가능합니다. 본 기술을 이용한 유전체 수준에서의 타겟 프로파일을 분석 시 독성 약물은 비독성 약물에 비해 폭 넓은 타겟 스펙트럼을 보여주었습니다. 이는 해당 약물의 인체에서의 독성 양태를 잘 대변하고 있으며, GPScreen™ 기술을 이용 시 동종의 신약 개발 후보 물질들 중에서 보다 안전한 약물을 선별 (drug prioritization) 함에 있어 매우 효과적으로 사용 가능합니다.
Figure 3. 독성 및 비독성 약물을 각각 GPScreen™ 한 후 타겟 스펙트럼 비교